5月4日,学校生物学研究中心王翊教授课题组在生物领域国际顶级期刊《Nucleic Acids Research》(《核酸研究》,IF: 11.561)在线发表了题为“OrthoVenn2: a web server for whole-genome comparison and annotation of orthologous clusters across multiple species”(OrthoVenn2:一个用于多物种全基因组直系同源基因簇比较和注释的在线工具)的原创性研究论文,发布了用于比较和分析全基因组直系同源基因簇并生成可视化结果的在线工具。
同源基因可分为两大类:直系同源基因和旁系同源基因。直系同源基因起源于共同的祖先,如果物种间的直系同源基因显示高度的序列相似性,则它们可能具有相似的生物学功能。另一方面,在进化分歧较大的物种间的直系同源基因可能发挥不同的功能。近年来,鉴定直系同源基因并确定它们之间的相似程度是比较基因组学研究中理解基因和基因组进化的重要内容。目前已经开发了两种用于鉴定直系同源基因的基本方法。第一种是基于树的,如PhyloTreePruner和TreeFam等工具。第二种是基于图形的,如PanOCT,OrthoFinder,OrthoMCL,OrthoDB,OMA和Roary等工具。基于树和基于图的算法在许多方面都不同,并且每个算法都有其自身的优势。然而这些工具基本基于Linux系统,对其他操作系统用户不友好,缺乏可视化功能,对结果展示不够清晰明了。
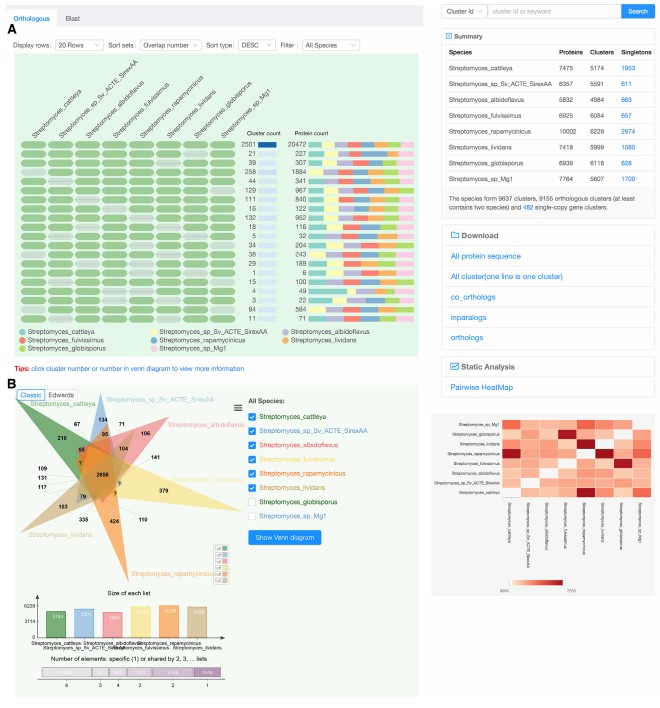
在论文发表之前,王翊教授已发布在线工具OrthoVenn,四年内引用接近200,该工具受到超过60多个国家和地区的研究者们使用。此次针对更广泛的比较基因组学研究人员的需求,王翊课题组将OrthoVenn全面升级为OrthoVenn2。课题组从Ensembl数据库下载了多物种的蛋白质序列并整合到OrthoVenn2中。OrthoVenn2使用OrthoMCL提供的启发式最佳匹配方法来识别基于保守的直系同源基因,允许用户对多达十二种不同物种进行快速全基因组比较并注释直系同源基因簇,这些物种包括植物,脊椎动物,细菌,真菌,原生生物和后生动物。用户还能提交自己的蛋白质序列给OrthoVenn2,在短时间内就能得到全面的同源基因比较和注释的可视化结果。OrthoVenn2采用HTML5和JavaScript全面提升了分析结果的可视化程度,使用维恩图和聚类表来显示多个物种的直系同源聚类分布。同时还提供功能注释,多序列比对,蛋白质互作网络,基序分析和系统发育树等结果展示。OrthoVenn2还基于Docker开发了本地版本,方便用户下载使用,本地版对不同操作系统用户提供了友好且丰富的可视化结果,且没有物种数量限制,方便用户布置到任意服务器上,对直系同源基因的研究具有重要意义。
西南大学为该论文成果的第一完成单位,王翊教授为论文的唯一通讯作者。文章同时致谢前沿交叉学科研究院生物学研究中心夏庆友教授、生物大数据分析团队的开发小组(鲁方,罗勇江)、文档小组(徐凌,董照斌)、测试小组(魏昭渊,郭海龙,张国庆)。该项研究得到了国家自然科学基金委面上项目、西南大学生物学“双一流”建设项目以及“聚贤工程”人才启动项目等基金资助。王翊教授还受到国家海外高层次人才青年项目以及重庆市留学生创新创业计划项目支持。
原文链接:https://academic.oup.com/nar/advance-article/doi/10.1093/nar/gkz333/5485531
(前沿交叉学科研究院生物学研究中心供稿供图 编辑 韩笑 审核 周翱)